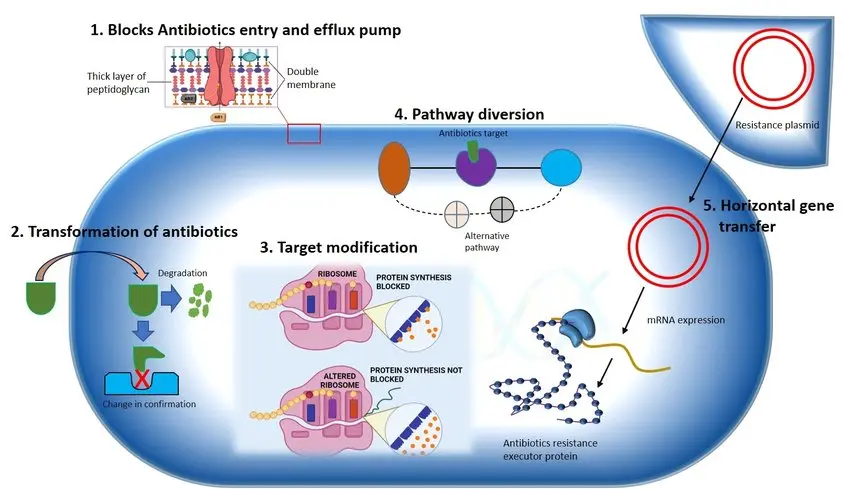
Metagenomische Studien haben gezeigt, dass Antibiotikaresistenzgene in der Umwelt allgegenwärtig sind. Dies lässt vermuten, dass ein hohes Risiko besteht, dass diese Gene auf Bakterien übertragen werden, die Infektionen beim Menschen verursachen. Sollte dies zutreffen, ist es sehr schwierig, das tatsächliche Risiko der Verbreitung von Resistenzgenen aus Umweltreservoirs auf menschliche Krankheitserreger abzuschätzen.

Enzymatische Strategien zur Inaktivierung von Antibiotika

Hydrolyse
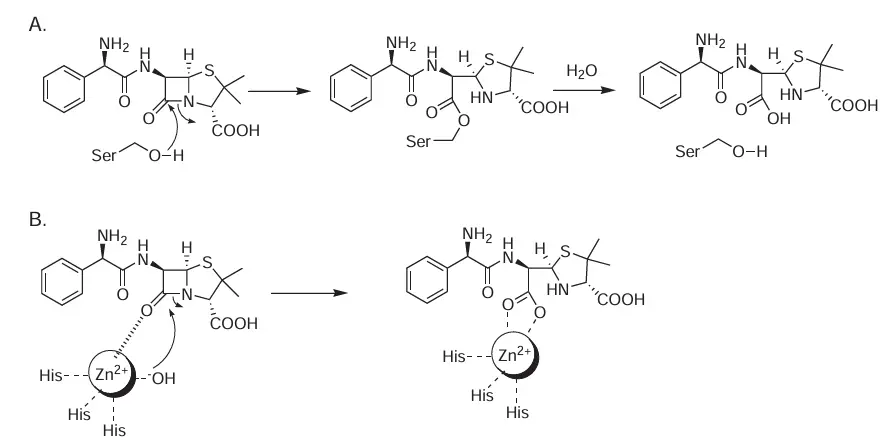
Eine der häufigsten enzymatischen Strategien von Bakterien zur Inaktivierung von Antibiotika ist die Hydrolyse – eine chemische Reaktion, die das antibiotische Molekül zersetzt und dadurch unwirksam macht. Dieser Mechanismus ist besonders bei β-Lactam-Antibiotika wie Penicillinen und Cephalosporinen bekannt. Bakterien produzieren β-Lactamase-Enzyme, die den für die antibakterielle Wirkung des Antibiotikums essentiellen β-Lactam-Ring hydrolysieren. Ist dieser Ring geöffnet, kann das Medikament nicht mehr an sein Ziel, die Enzyme der bakteriellen Zellwandsynthese, binden, und das Bakterium überlebt. Die Gene für β-Lactamasen können auf Plasmiden gespeichert werden, was eine schnelle Ausbreitung zwischen Bakterien durch horizontalen Gentransfer ermöglicht. Das weit verbreitete Vorkommen hydrolytischer Enzyme wie Extended-Spectrum-β-Lactamasen (ESBLs) und Carbapenemasen stellt eine erhebliche Herausforderung für die klinische Behandlung dar und erfordert neue Antibiotika und β-Lactamase-Hemmer, um diesem Resistenzmechanismus entgegenzuwirken.
Gruppentransfer
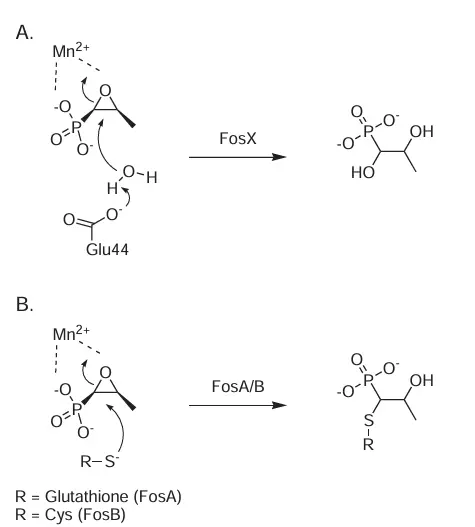
Die vielfältigste und damit größte Familie von Resistenzenzymen sind die Gruppentransferasen. Diese Enzyme inaktivieren Antibiotika durch kovalente Modifikation. Dies führt zu strukturellen Veränderungen, die die Bindungsfähigkeit des Antibiotikums an sein Ziel verringern oder blockieren. Gängige chemische Strategien der Gruppentransferasen sind O- und N-Acylierung, O-Phosphorylierung, O-Nukleotidylierung, O-Ribosylierung, O-Glykosylierung und Thioltransfer. Alle diese kovalenten Modifikationsmechanismen benötigen für ihre Aktivität ein Cosubstrat wie ATP, Acetyl-CoA, NAD+, UDP-Glucose oder Glutathion. Da diese Cosubstrate zellintern vorhanden sind, wirken diese Enzyme ausschließlich im Zytosol.
Andere enzymatische Resistenzmechanismen
Redox-Enzyme
Redoxenzyme stellen eine weitere Klasse bakterieller Resistenzmechanismen dar, sind jedoch weniger verbreitet als hydrolytische Enzyme oder Transferasen. Diese Enzyme inaktivieren Antibiotika, indem sie Oxidations-Reduktions-Reaktionen (Redoxreaktionen) katalysieren, die die Struktur des Antibiotikums chemisch verändern und dabei oft wichtige funktionelle Gruppen zerstören, die für die antimikrobielle Aktivität erforderlich sind. Beispielsweise können bestimmte Bakterien Reduktasen produzieren, die nitroaromatische Antibiotika wie Nitroimidazole durch Reduzierung ihrer Nitrogruppen inaktivieren. Ebenso können oxidative Enzyme Antibiotika wie Aminoglykoside modifizieren und ihre Wirksamkeit verringern. Anders als viele Resistenzenzyme, die auf kovalenter Modifikation oder Hydrolyse beruhen, sind Redoxenzyme auf Elektronentransferreaktionen angewiesen und benötigen oft Kofaktoren wie NADH oder NADPH. Obwohl sie nicht so weit verbreitet ist wie andere Resistenzstrategien, unterstreicht die redoxbasierte Inaktivierung die bemerkenswerte biochemische Vielfalt, die Bakterien nutzen können, um Antibiotikastress zu überleben.
Lyasen
Lyasen sind eine relativ seltene, aber bemerkenswerte Enzymklasse, die durch die nicht-hydrolytische Spaltung chemischer Bindungen in Antibiotikamolekülen zur Antibiotikaresistenz beitragen kann. Im Gegensatz zu Hydrolasen, die Wasser zum Aufbrechen von Bindungen verwenden, entfernen Lyasen funktionelle Gruppen durch Mechanismen wie Eliminations- oder Additionsreaktionen, was zu strukturellen Veränderungen führt, die das Antibiotikum inaktivieren. Beispielsweise wurde gezeigt, dass einige Bakterien lyaseähnliche Enzyme produzieren, die bestimmte zyklische Strukturen in Antibiotikamolekülen spalten und so deren Bindungsfähigkeit an bakterielle Ziele beeinträchtigen. Obwohl Lyase-vermittelte Resistenzen nicht so häufig oder weit verbreitet sind wie Hydrolyse- oder Gruppentransferase-Strategien, verdeutlichen sie die bemerkenswerte biochemische Anpassungsfähigkeit von Bakterien und unterstreichen die Notwendigkeit einer kontinuierlichen Überwachung neu auftretender Resistenzmechanismen.
Effluxpumpen


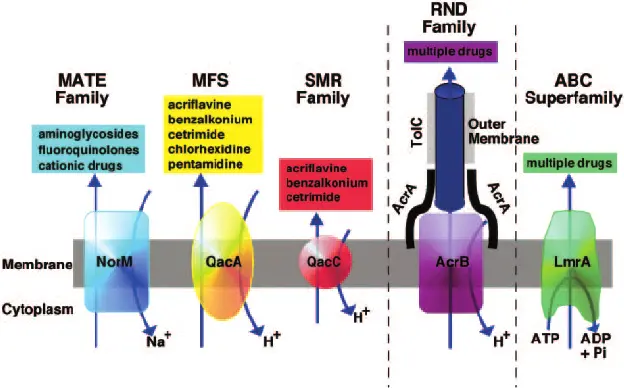
Effluxpumpen sind einer der wichtigsten und am weitesten verbreiteten Mechanismen, mit denen Bakterien Resistenzen gegen Antibiotika aufbauen. Diese Membranproteine transportieren aktiv toxische Substanzen, darunter auch Antibiotika, aus der Bakterienzelle und senken so die intrazelluläre Konzentration des Medikaments auf subletale Werte. Effluxpumpen können spezifisch für ein einzelnes Antibiotikum oder ein Breitbandantibiotikum sein und mehrere Medikamentenklassen ausschließen, was zur Multiresistenz (MDR) beiträgt. Es gibt verschiedene Familien von Effluxpumpen, wie die Major-Facilitator-Superfamilie (MFS), die Resistance-Nodulation-Division-Familie (RND) und die ATP-Bindungskassetten-Transporter (ABC). Viele Effluxpumpengene befinden sich auf Chromosomen, können aber auch über Plasmide aufgenommen werden und sich so unter Bakterien verbreiten. Eine Überexpression dieser Pumpen, die oft durch Umweltstress oder Antibiotikaexposition ausgelöst wird, kann die Wirksamkeit einer Behandlung erheblich beeinträchtigen und ist ein großes Problem in der klinischen Mikrobiologie.

Zielmodifikation


Umgehungswege

Bypass-Wege sind eine clevere Resistenzstrategie, die es Bakterien ermöglicht, eine Antibiotikabehandlung zu überleben, indem sie den blockierten Stoffwechselschritt, auf den das Medikament abzielt, umgehen. Statt sich lediglich auf die Modifizierung des Medikaments oder seines Ziels zu verlassen, entwickeln Bakterien alternative biochemische Wege, um wichtige Zellfunktionen auszuführen. Ein klassisches Beispiel ist die Sulfonamid- und Trimethoprim-Resistenz: Diese Antibiotika hemmen die Folsäuresynthese, indem sie spezifische Enzyme des Stoffwechselwegs blockieren. Manche Bakterien eignen sich jedoch neue Enzyme mit geringerer Affinität zum Medikament an oder können Folat direkt aus der Umgebung importieren, wodurch der gehemmte Schritt effektiv umgangen wird. Ein weiteres Beispiel ist die Aufnahme alternativer Penicillin-bindender Proteine (PBPs) bei manchen Bakterien, die trotz β-Lactam-Antibiotika die Zellwandsynthese fortsetzen. Durch die Nutzung von Bypass-Wegen können Bakterien überleben und wachsen, selbst wenn der primäre Stoffwechselweg durch die Wirkung von Antibiotika unterbrochen wird, wodurch Infektionen schwerer auszurotten sind.